選択的スプライシングとは、未成熟mRNAのどの領域をエクソンとするか(逆に言うとイントロンとしてどこを切り出すか)を変えることにより、1つの遺伝子から塩基配列の異なる複数種類の成熟mRNAを作り出す機構です。
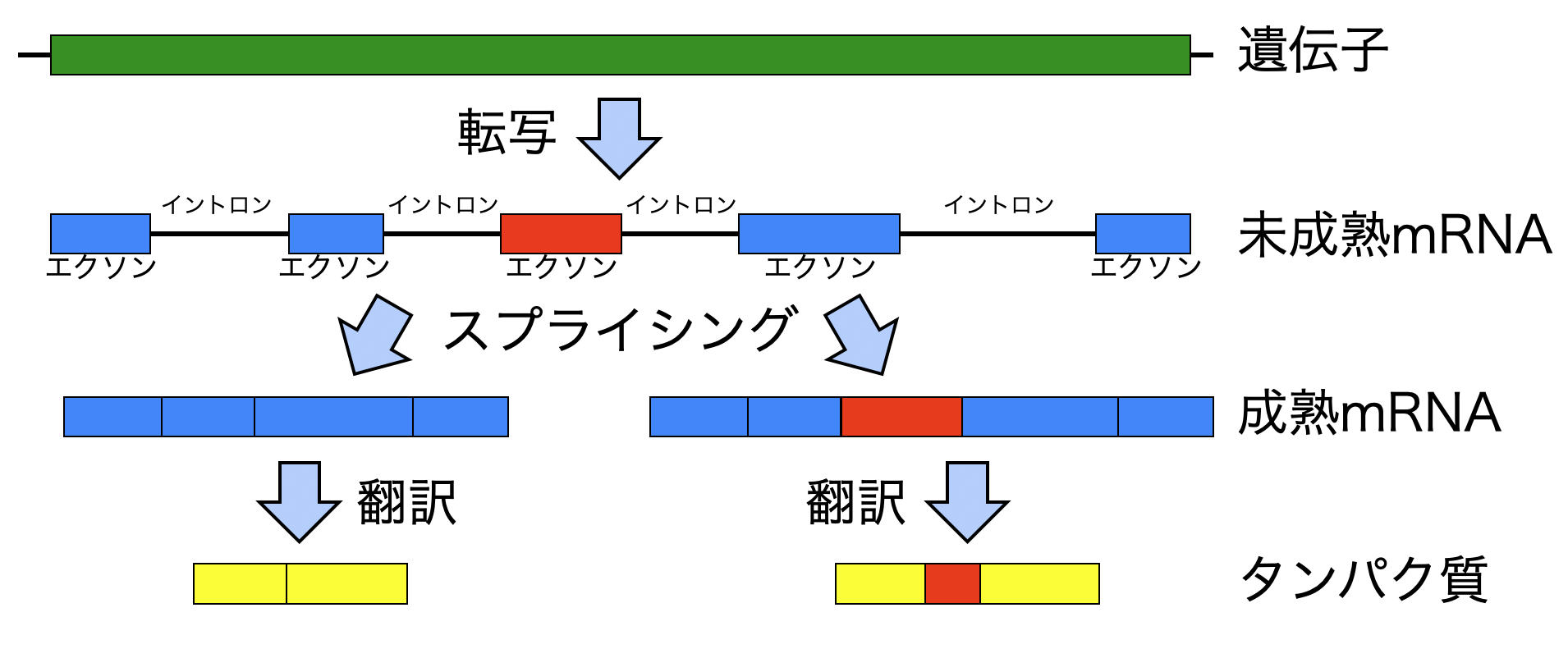
ヒトゲノムが決定した時に、予想以上に遺伝子数が少なく、線虫やハエに比べて体の複雑さの割に遺伝子数の差が小さいことが明らかとなりました。同時期に、それまではごく限られた遺伝子だけで見られる例外的な現象だと思われていた選択的スプライシングが、非常に多くの遺伝子で起こっていることも明らかとなり、高等真核生物では、遺伝子数が少なくても、選択的スプライシングが起きることにより生物学的な複雑性が生じているという考え方が一般的となりました。
つまり、遺伝子が少なくても、mRNAの転写の後にイントロンの場所を変えてやれば遺伝子が多数あることと同じような効果を示すという考え方です。
この説は非常に魅力的ですが、少し疑問が残ります。選択的スプライシングによりいろいろな種類のものが作られるのはあくまでも成熟mRNAです。大部分の遺伝子は、タンパク質に翻訳されて初めて機能するため、いろいろな種類の成熟mRNAが翻訳され、遺伝子がたくさんあるのと同様に異なる機能を果たしているかは保証されません。
そこで、選択的スプライシングによりできる成熟mRNAがどんなタンパク質をコードしているのか?、また、そのタンパク質はどんな機能を果たしそうなのかを解析していくことを興味として研究を行っています。
具体的には、ヒトの選択的スプライシングにより作られるタンパク質(スプライシングアイソフォーム)のうち、よく知られているアイソフォーム(メジャーアイソフォーム)のどのような場所が、あまり知られていないアイソフォーム(マイナーアイソフォーム)で変化するかを解析しました。その結果、メジャーアイソフォームにおいてタンパク質の構造形成に重要な疎水性コアを形成している部分や、機能に直接関わる部位がマイナーアイソフォームで失われていたり大きく変化していたりする場合が多いことが明らかとなりました(Yura et al. 2006)。疎水性コアの一部が欠失しているマイナーアイソフォームでは、タンパク質が機能するのに重要な立体構造が不安定化すると考えられ、機能部位そのものを欠失することも含めて、選択的スプライシングがタンパク質の機能を壊すスプライシングアイソフォームを多く作っていると考えられました。
ヒトやマウスなどのモデル生物のスプライシングアイソフォームを網羅的に集めたデータベースであるAS-ALPSを構築し(Shionyu et al. 2009)、同様の解析を行っても、やはり選択的スプライシングがタンパク質の機能を壊すスプライシングアイソフォームを多く作り出しているという結果が得られています。
では、疎水性コアの一部が欠失したマイナーアイソフォームは本当に立体構造が不安定化しているのでしょうか。そのことを確認するため、タンパク質の立体構造を精度良く予測できるAlphaFoldを使って構造予測を行いました。
例えば、解糖系を構成するタンパク質であるβエノラーゼの場合、マイナーアイソフォームの予測構造は、タンパク質の主鎖構造だけを見ると一見普通の構造に見えますが、実際は大きな穴があいていて疎水性のアミノ酸が溶媒に露出する非常に不自然な構造が予測されることがわかりました。
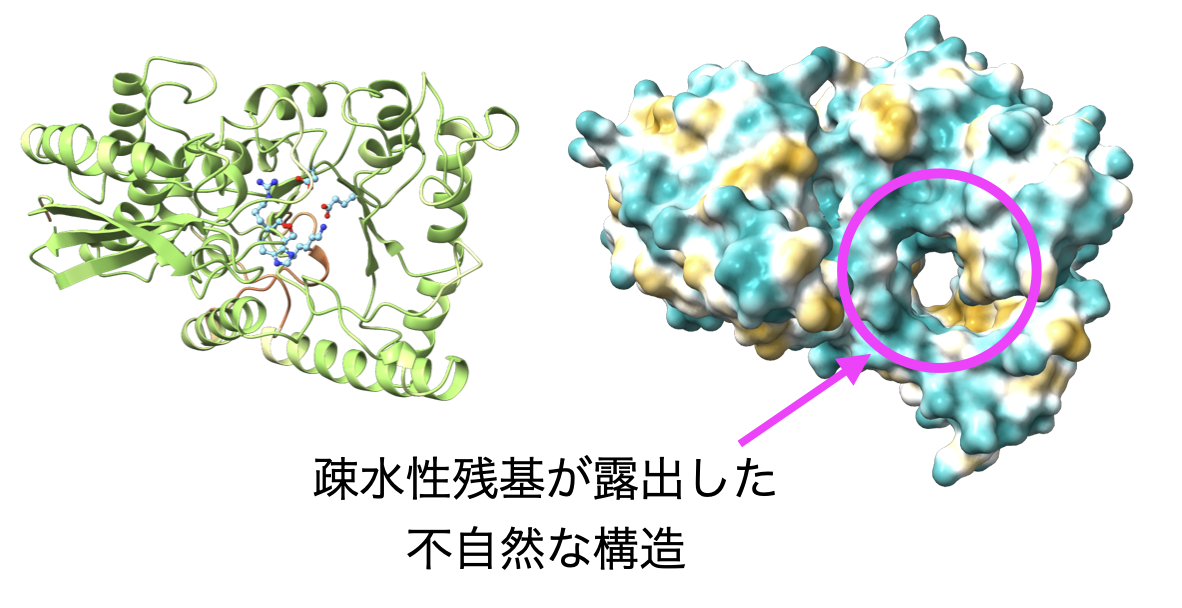
AlphaFoldが間違った立体構造を予測しているのか、それとも実際に生体内ではこのような構造のマイナーアイソフォームが作られ、何らかの機能を持っているのかはまだわかりません。今後、このような予測構造の評価や機能予測に取り組んでいきます。