補酵素結合予測法に基づく新規酵素の探索
ゲノムの塩基配列が決定されると、それにコードされるタンパク質のアミノ酸配列情報を網羅的に求めることができます。一般的には、得られたタンパク質がどのような機能を持つかを推定するために既知のタンパク質に対して相同性検索を行い、機能がわかっている似たタンパク質がないかを探します。
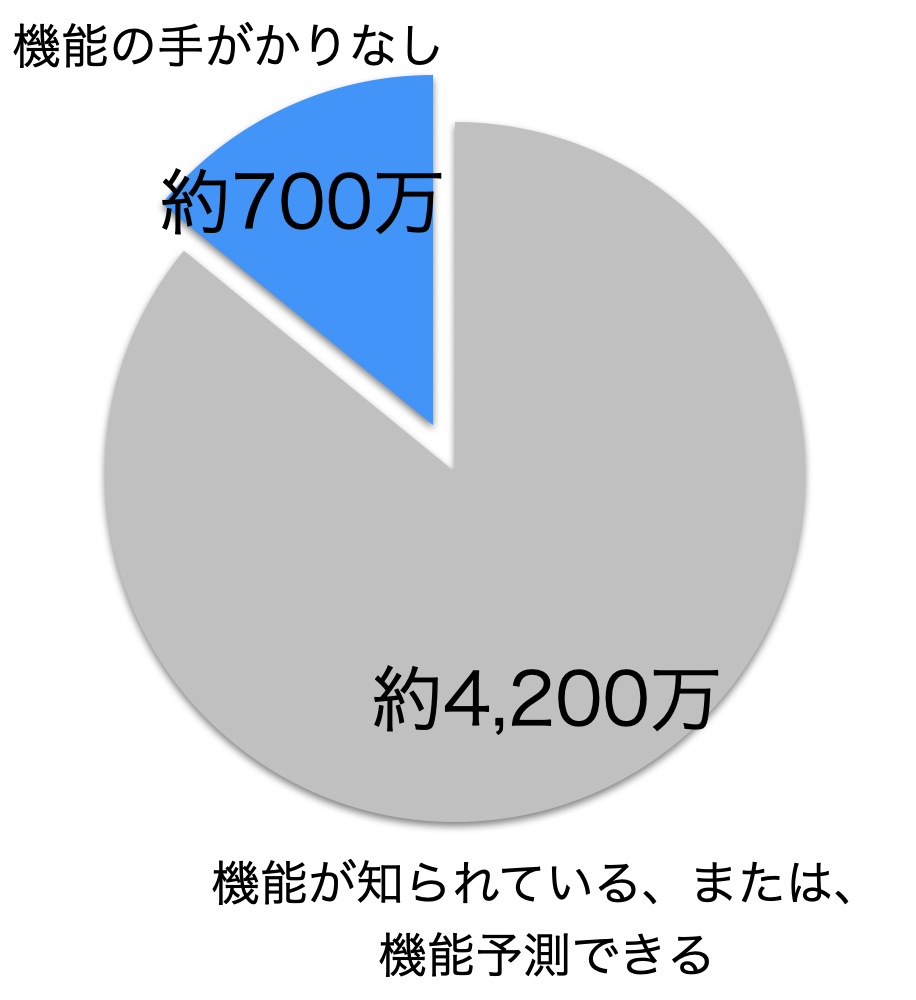
このようにして多くのタンパク質の機能が推定されていますが、既知のタンパク質アミノ酸配列のうち、15%以上は機能の手がかりが得られていません。生物が持っている代謝経路から考えると存在するはずの酵素が見つからないという場合があり、機能がわからないタンパク質の一部はこのような未知の酵素である可能性があります。
我々は配列の相同性に依存しない形で酵素活性に重要な補酵素の結合を予測する方法を開発することで、新規の酵素を探索することを目指しています。
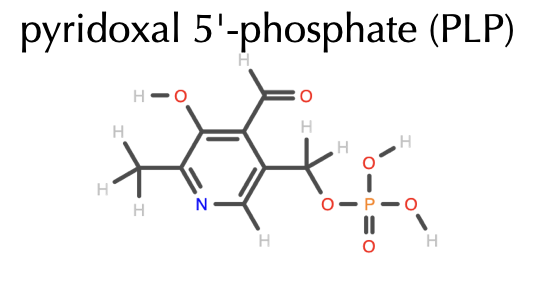
その手始めとして、アミノ酸代謝に関わる酵素でよく使われる補酵素であるピリドキサールリン酸(PLP)が結合するタンパク質を対象として予測法を開発しました。
この予測法では、既知のタンパク質とリガンドの複合体情報をデータベース化したHet-PDB Navi2から得られるデータを活用し、既知のPLPが結合するタンパク質の立体構造の特徴をグラフニューラルネットワークを使って学習することで、PLP結合に関わるアミノ酸残基やPLPと結合するかを予測できるようにしています。
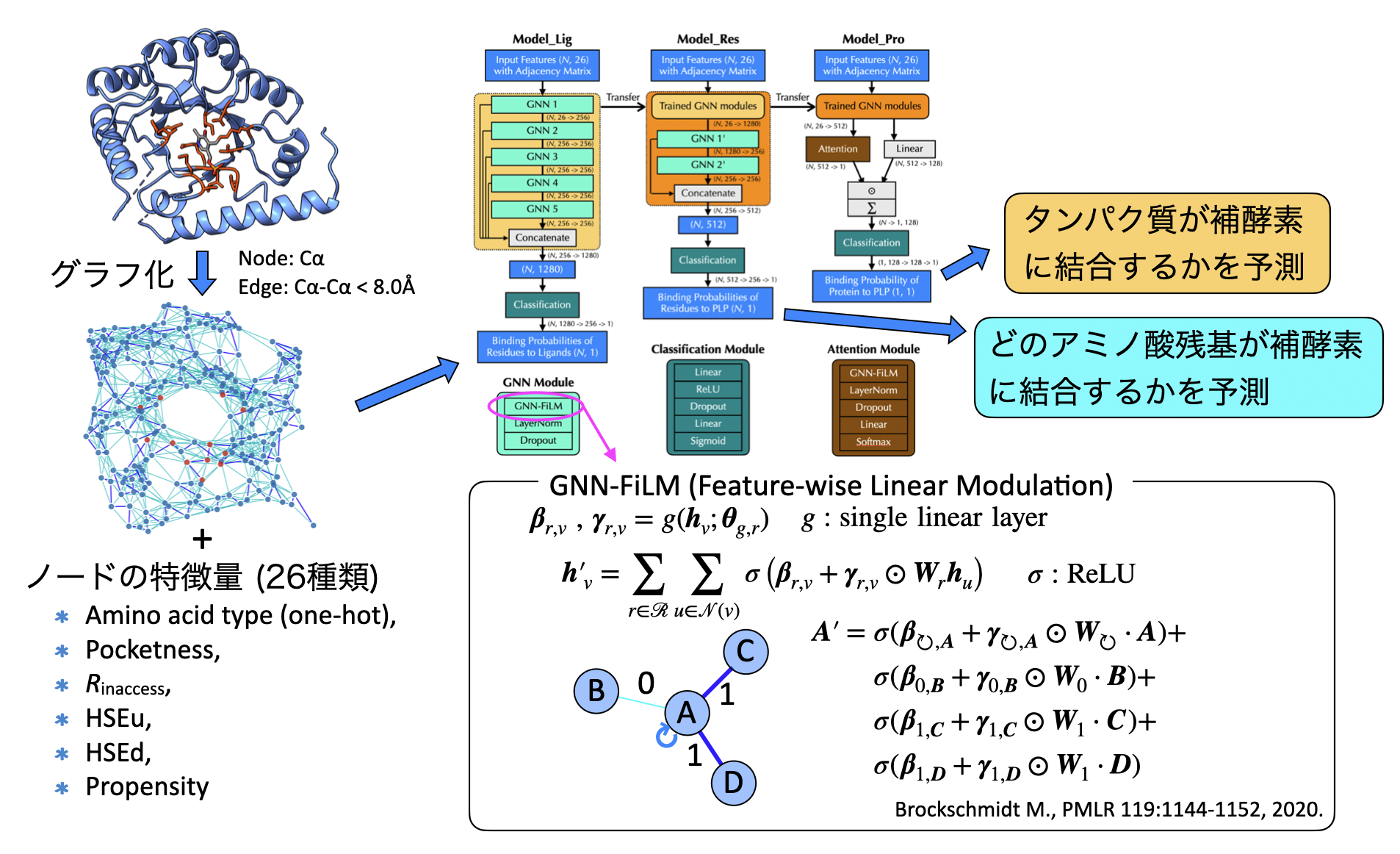
現在は、出芽酵母において機能がわかっていないタンパク質に対してPLPの結合予測を行い、本学の応用微生物学研究室、分子生物化学研究室と共同で、実際にPLPと結合するかどうかを検証を行っています。
ドッキングによるタンパク質-リガンド複合体構造の予測
近年、X線結晶解析やクライオ電子顕微鏡などのタンパク質立体構造の決定手法が発達しており、非常に多数の立体構造情報が蓄積しています。また、DeepMind社によるAlphaFoldをはじめとする深層学習を用いた高精度立体構造予測法が普及したことにより、さらに多くのタンパク質について立体構造情報が得られるようになりました。
タンパク質は多くの場合、他の分子(リガンド)と結合することで機能するため、タンパク質とリガンドの構造は機能を理解する上で非常に重要です。しかし、決定/予測された立体構造にそのようなリガンドが含まれていない場合も多くあります。
リガンドとの結合構造を予測する方法としてリガンドドッキングがあります。リガンドドッキングでは、リガンドが取りうる構造を探索しながら、リガンドが結合するタンパク質の表面構造およびその化学的性質とできるだけ相補的になるようタンパク質-リガンド複合体構造を求めます。
塩生研では、本学の構造生物学研究室との共同研究において、新型コロナウイルス感染症の治療薬候補がその標的と考えられるタンパク質とどのように結合するかを報告しています(Hijikata et al. 2022)。また、奈良先端大学院大学および本学の構造生物学研究室との共同研究ではROS代謝酵素とクルクミン誘導体の複合体構造(Lestari et al. 2019, Nakamae et al. 2019)をドッキングにより予測した結果を報告しました。他にも、本学の蛋白質機能解析学研究室との共同研究においてヒト20Sプロテアソームとその阻害剤の複合体構造(Hasegawa et al. 2014, Tanaka et al. 2018)や、ヒト20Sプロテアソームとそのタンパク質分解活性を上昇させる人工進化ペプチドとの複合体構造(Zhu Y et al. 2023)、バクテリアのPseudomonas fulvaが持つピオベルジンの合成酵素とその基質の複合体構造(Ochiai et al. 2025)など、様々なタンパク質においてリガンドとの複合体構造を予測しています。