ヒトゲノムの特徴
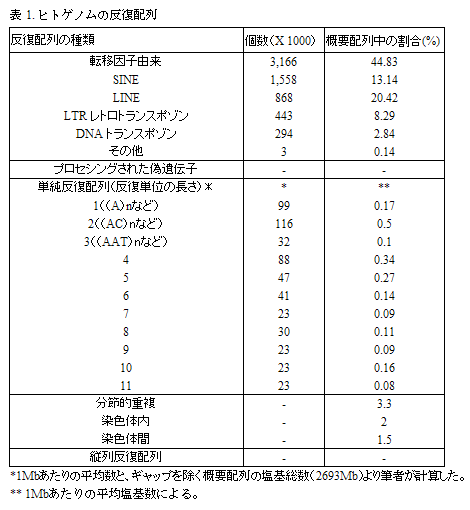 |
膨大な反復配列の存在は、哺乳類ゲノムの解読を困難にしている要因の一つです。反復配列は配列決定の障害であり、またゲノムの種間比較や、コンピュータによる機能領域の予測を一層困難なものにしています。このため現在では、ゲノム情報の処理には、まずコンピュータプログラムにより反復配列を隠すのが定石になっています1)2)。しかし、反復配列は本当にわれわれのゲノムにとって有害無益なだけの存在なのでしょうか。 ヒトのタンパク質コード配列はゲノムの5%に満たないが、反復配列は少なくとも50%を占めており、変異で識別が困難になったものも含めればそれよりも多いと考えられています。これらの反復配列は5種類に大別することができます[表1]3)。 このページでは、初めにヒトゲノム概要配列の解析結果基づき、哺乳類ゲノムに極めて豊富に存在する、トランスポゾンに由来する反復配列について解説します。次いでヒト遺伝子のゲノム内での分布に関する話題を述べ、最後に、プロセシング済み偽遺伝子に関する我々の解析結果を紹介します (研究成果:総説その他3を一部改訂して掲載)。 |
|---|
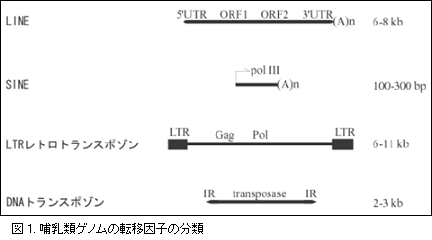 |
哺乳類の転移因子は、長い散在性因子(LINE)、短い散在性因子(SINE)、LTRレトロトランスポゾン、あるいはDNAトランスポゾンのいずれかに分類されます[図1]。前3種類はRNA中間体を介して転移し、最後のものは直接DNAとして転移します。RepeatMaskerプログラム1)で識別可能なSINE、LINE、LTRレトロトランスポゾンおよびDNAトランスポゾンのコピーは、概要配列の各々13%、20%、8%および3%を占めます[表1]3)。つまり、ヒトゲノムのおよそ45%が、このような転移因子に由来しています。古い(非常に変異した)反復配列を含めれば、この数値はさらに大きくなるでしょう。 ヒトゲノムに存在する反復配列の多くは、過去に転移活性のあった転移因子に由来しています。転移因子のコピーは、ゲノムへ挿入した時点から現在までの期間に、ランダムな塩基置換を蓄積しています。中立的な塩基の置換数は時間の経過とともに増加するため、祖先転移因子とそのコピーの塩基配列相違度を求めれば、そのコピーがゲノムに生じた実年代を概算することができます。 ヒト概要配列中の散在性反復配列の各々について、このような解析がなされ、いくつかの事実が明らかになっています3)。まず、ヒトゲノム散在性反復配列の多くは、真獣類(カモノハシやカンガルーなどの原始的哺乳類を除く哺乳類)の放散(6500万年~9000万年前)よりも前に生じています。LINEとSINEは非常に長い寿命を持っており、L1とAluの一部は、各々少なくとも1億5000万年および8000万年前から存在しています。初期の哺乳類ゲノムでは、LINE2とMIRの転移が活発であったが、8000万年から1億年前にはそれらの転移活性は著しく減少し、やがて消失しました。 過去5000万年の間に、ヒトゲノムでDNAトランスポゾンが活動した証拠は見出されておらず、LTRレトロトランスポゾンも、現在では転移活性が低いです。しかし、マウスではIAPと呼ばれるLTRレトロトランスポゾンが現在でも活発に転移しています。ヒト科の系統では転移因子の活動は、全般的に過去3500~5000万年間衰退傾向にありますが、約4000万年前には例外的に、Aluの爆発的増幅が生じています。この問題には、後半の偽遺伝子の節で再び触れます。 |
|---|
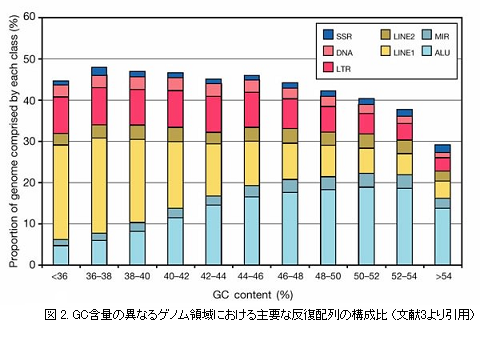 |
LINE配列はヒトゲノムの中でもGC含量が低い領域(ATに富む領域)に豊富に存在していますが、SINE(MIR,Alu)はこれとは逆に、GC含量の高い領域に豊富に存在しています。一方、LTRレトロトランスポゾンやDNAトランスポゾンは、異なるGC含量の領域にほぼ一様に分布しています[図2]3)。L1がATに富む領域に豊富なのは、L1のエンドヌクレアーゼがATに富む塩基配列(TTTTA)を標的としてそのDNAを開裂することが原因かもしれません4)。しかし、L1の転移機構5)6)に依存するAluが正反対の分布を示すのは、実に奇妙な現象です。 Aluを生成年代順に4つのグループに分け、各々のゲノム内分布を調べた結果、最近のAluはL1と同様にATに富む領域に豊富であるが、古いAluほど、GC含量の高い領域に豊富であることが示されました[図3]3)。過去3000万年程度の期間で、Alu はGC含量の高い領域で、実に13倍に濃縮されたことになります。 |
|---|---|
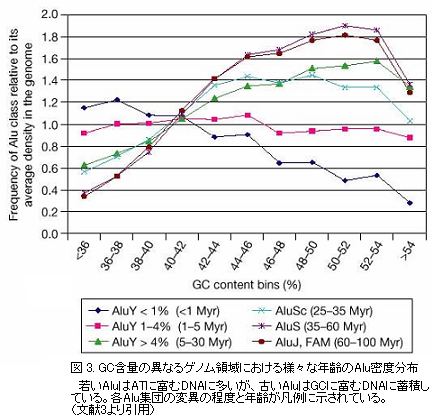 |
どのような原因でAluの急速な濃縮が生じたのでしょうか。次の節で述べるように、GC含量の高い領域には遺伝子が豊富です。このためGC含量の高い領域よりもATに富む領域のAluの方が、欠失し易い可能性も考えられます。しかしながら、L1では過去1億年に渡って、Aluのような顕著な分布変化が見られないため、この可能性は考えにくいです。GC含量の高い領域のAluに対する正の選択(進化的な圧力)が存在するのではないか、というのが概要配列解析論文の論調です。Alu の生物学的機能を示唆する報告もあります。Aluからの転写産物RNAは、生体のストレス応答に際してタンパク質合成制御に貢献しているかもしれません7)。 SINEとLINEのこのように偏向したゲノム内分布は、マウスでも報告されています8)。ヒトとマウスの系統で、各々独立にゲノムに挿入したSINEやLINEのゲノム内分布を調べた結果、両種のSINE分布には極めて高い相関が見られました。一方のゲノム内でのSINE分布から、他方のSINEの分布が予測できる程、その相関は高いものでした。 転移因子のゲノム内での不均一な分布が、転移因子の転移メカニズムや、DNA組み換えなどのゲノムの動的変動に関連したものなのか、あるいは、Aluなど一部の反復配列の生物学的機能に関連したものであるのか、今後解明されるべき課題でしょう。 |
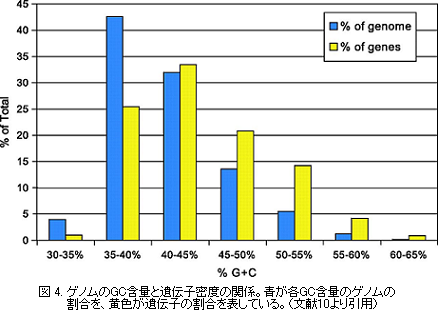 |
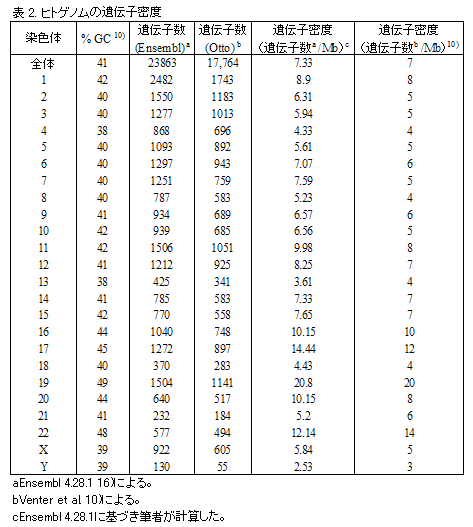
ゲノム内の遺伝子分布を表現する時、砂漠に遺伝子のオアシスが点在するという比喩がよく用いられます。実際、ゲノムの20%が500kb以上にわたって遺伝子が存在しない砂漠です9)10)。遺伝子はGCに富んだDNA領域に多く、GCに乏しいDNA領域には少ない傾向にあります3)10)。50kbのスケールでは、GC含量48%以上のゲノム領域は全体の9.5%ですが、そこに存在する遺伝子は全体の24.8%であり、一方GC含量43%未満のゲノム領域は全体の69.2%ですが、遺伝子は48.5%です[図4]。 染色体上の遺伝子数は、概ね各染色体の真性クロマチン領域の大きさに比例しています。しかしこの相関は厳密なものではありません。単位塩基数あたりの遺伝子数を染色体ごとに計算すると、その数値は1Mbあたり3個~20個3)10)と、染色体の間で大きく異なっていることがわかります[表2]。このような基準で判断すると、19、17番染色体は「遺伝子に富んだ」染色体、Y、13、4番染色体は「遺伝子に乏しい」染色体であるということができます。染色体遺伝子密度は、染色体全体の平均GC含量とよい相関があります10)。遺伝子密度は、染色体規模のスケールでもDNAのGC含量と深い関係にあるようです。DNAのGC含量は、DNAの複製時期や修復機構と密接に関わっていることから11)、GC含量に対応した染色体上の遺伝子分布の偏りには、染色体の構築に関係した生物学的意義が存在するのではないでしょうか。 |
|---|---|
 |
ヒトとチンパンジーのような近縁な生物では、ゲノム上の各遺伝子の順列はほぼ等しいです。しかし、ヒトとマウス、ヒトとフグというように、系統関係が隔たるにつれて、次第にそれらの相対的配置はばらばらになっていきます。この現象は、進化の過程におけるゲノムの巨視的な変化、すなわち転座、逆位、融合といった染色体レベルの変異が原因であると考えられます。 興味深いのは、ヒトとフグなどの遠縁の生物ゲノムを比較しても、遺伝子の並び(シンテニー)が保存されたDNA領域(シンテニック領域)を見出せることです12)。保存されたシンテニーの多くは、染色体の再編を免れた偶然の産物かもしれませんが、中には、機能的な制約から特定の遺伝子配置が保存されている例もあります。胚の体軸に沿った発現パターンを示すHox遺伝子群などは、遺伝子の発現パターンに対応して染色体上に順に配置しています13)14)。今後、保存されたシンテニーの一部から、それら遺伝子群の機能的な関連性が発見される可能性があるかもしれません。 |
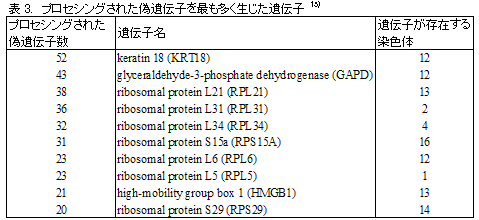 |
ゲノムには、遺伝子と類似した塩基配列であるが機能は持たないと推測される配列が多数存在しています。このような配列を偽遺伝子と呼びます。偽遺伝子は生成機構から2種類に大別することができます。「DNA重複型偽遺伝子」は、不等交叉などの結果生じた遺伝子の重複構造が、塩基置換等の変異により機能を失ったものです。「プロセシング済み偽遺伝子」は、細胞のmRNAが逆転写反応によりcDNA化し、ゲノムに再挿入されて生じたものです。後者は一般に、プロモーター配列を失うために、生成直後から転写活性を失います。 ヒトを含む哺乳類のゲノムには、プロセシング済み偽遺伝子が極めて多いことが以前から知られていました。最近、我々はプロセシング済み偽遺伝子をヒトゲノムから網羅的に抽出し、解析をおこなったので、ここではその概要を紹介したいと思います15)。 ヒト遺伝子の23,929種類の転写産物16)を用いて、ヒトゲノム配列から、プロセシング済み偽遺伝子の候補配列を抽出しました。1,299種類の転写産物に対する3,664個のプロセシング済み偽遺伝子配列を得て、それらの解析をおこないました。最も多くの偽遺伝子が検出された遺伝子はkeratin 18 (KRT18)(52個)であり、次いでglyceraldehyde-3-phosphate dehydrogenase (GAPD)(43個)、ribosomal protein L21 (RPL21)(38個)の順でした[表3]。 解析した諸性質の中でも特に興味深い結果は、プロセシング済み偽遺伝子の生成年代に関するものです。各々のプロセシング済み偽遺伝子の塩基置換の程度から、生成年代を推定し、生成年代を横軸にとったヒストグラムを作成したところ、その分布がポアソン型(一つの頂点を持った山型)を示しました。この事実は、霊長類進化のある時期ヒトの祖先のゲノムで、プロセシング済み偽遺伝子が爆発的に増幅した可能性を示唆しています。そのピークの時期は4000-5000万年前と推定されました。 |
|---|---|
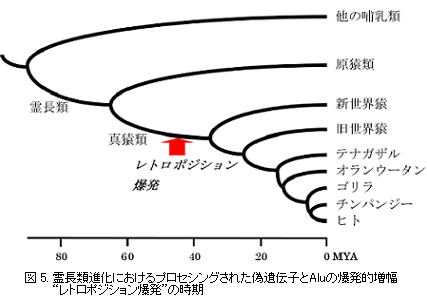 |
興味深いことに、ちょうどこの時期、Alu (ヒトゲノムの10%を占めるSINE)にも同様の爆発的増幅が生じています。先にも紹介したように、AluがGC含量の高いゲノム領域に高密度に分布していることから、Aluには進化的な選択圧がかかっている可能性が議論されています。このため、Aluの爆発的増幅はAluの機能に関係しているのか、あるいは無機能な大部分のAluを生産する機構の変化に原因があるのか、Alu単独で考えてみても判然としません。しかし我々の結果は、プロセシング済み偽遺伝子とAluという全く別の転移因子が、霊長類進化の同時期に爆発的に増幅した可能性を示しています。このことから、当時、これら異種の転移因子に共通の転移機構に何らかの変化が生じ、この爆発的増幅の誘因となった可能性が浮上してきました[図5]。 プロセシング済み偽遺伝子やAlu の増幅は、LINE1(L1)の逆転写酵素に依存していると考えられています17)。それでは、L1もこの時期に同様の爆発的増幅を生じているのでしょうか。この問題を調べるため、次にヒトゲノムに存在する全てのL1の生成時期を再分析しました。L1の生成は、4000-5000万年前よりもさらに以前から、緩やかな減少の途上にあり、この時期だけの顕著な増幅は認められません。問題解明の鍵は、多種類存在するL1サブファミリーにあると思われます。 ヒトゲノムの全L1の生成時期を、約80種類のサブファミリー毎に分析した結果、L1PA6、L1PA7、L1PA8の各サブファミリーだけが、4000-5000万年前に集中的に転移していることが判明しました。このことから、プロセシング済み偽遺伝子とAlu の爆発的増幅には多くのサブファミリーの中でも、これらのサブファミリーが深く関与していると考えられます。現在のわれわれのゲノムで転移活性のあるL1(L1Hs)は、L1 RNA以外の細胞質RNAを逆転写する活性(トランス活性)が極めて低いです。先のサブファミリーにおいて、このトランス活性が一時的に亢進した結果、プロセシングされた偽遺伝子やAlu の爆発的増幅を生じたのではないでしょうか。 これらのサブファミリーはランダムな塩基置換を蓄積し、現在では転移活性を失い分子化石と化していますが、塩基配列を修繕することにより、かつての姿を復元することが可能です。培養細胞を用いたL1の転移アッセイ系と組み合わせれば、現在のL1との違いを検討することが可能でしょう。 |
| 本ページで紹介したように、ヒトを含めた高等真核生物のゲノムは、反復配列の存在により大きく変動しています。AluやL1の挿入やトリヌクレオチドリピートの伸長による遺伝性疾患などをみると、反復配列は生体やゲノムに負荷ばかり掛けているようにもみえます。しかし、これらはゲノム各所でむやみに増減を繰り返し、不要なゲノムを増築しているだけの存在ではないかもしれません。偽遺伝子の再活性化や、挿入配列の遺伝子発現制御への新規参入によって、生体構築情報系の革新やチューンアップに貢献しているかもしれません18)。過去にゲノムに組み込まれた反復配列と、現在の遺伝子発現制御ネットワークの関係を分析することにより、そのルールの一端を知る手掛かりが得られるのではないでしょうか。 |
|
